IMRAM
Institute of Multidisciplinary Research for Advanced Materials, Tohoku University
東北大学
多元物質科学研究所

LAST UPDATE 2023/08/07
-
研究者氏名
Researcher Name田口真彦 Masahiko TAGUCHI
助教 Assistant Professor -
所属
Professional Affiliation東北大学多元物質科学研究所
有機・生命科学研究部門 量子ビーム構造生物化学研究分野
Institute of Multidisciplinary Research for Advanced Materials, Tohoku University
Division of Organic- and Bio-materials Research, Quantum Beam-based Structural Biology and Chemistry -
研究キーワード
Research Keywords生体内化学反応
分子シミュレーション
量子/古典ハイブリッド計算法
Chemical reaction in biomolecular system
Molecular simulation
Hybrid QM/MM method
- 研究テーマ
Research Subject -
時分割実験と連携した計算科学による生体分子機能の理解と応用
Understanding of biomolecular function by computational science linked with time-resolved experiment and its application
研究の背景 Background
タンパク質分子の構造を原子レベルで時分割測定できる技術が開発され, その機能発現過程を直接視ることが可能となってきています. 一方で計算科学分野においても, タンパク質分子に特有な化学反応と遅い構造変化の両立した記述を可能にする方法論が開発されてきています. これらの技術を連携することにより, タンパク質分子の機能発現に伴う動的過程の理解が格段に進展することが期待されます.
Time-resolved measurement technique for structure of protein molecule at atomic level enables us to directly visualize functional expression process of protein molecule. On the other hands, in computational science field, a method compatibly describing chemical reaction and slow conformational change which are characteristic for protein molecule is now available. Linking the computational method with the time-resolved data expects us to deepen understanding for dynamical process of protein molecule upon its functional expression.
研究の目標 Outcome
時分割実験と連携し, タンパク質分子の機能発現に伴う動的過程を分子シミュレーションにより明らかにします. 反応プロファイルを補完するために, プロトン化状態等の化学構造の同定や遷移状態モデリングを行います. また, 機能発現に至るまでの化学反応に端を発する連鎖的な構造変化を捉えます. これらの研究を通して, タンパク質分子における構造と機能の関係性に対する理解を深め, “機能の多様性と進化による機能転換”に関する知見の獲得を目指します.
We clarify dynamical process of protein molecule upon its functional expression using molecular simulation linked with time-resolved measurement. To complement the chemical reaction profile, we identify the chemical structure such as protonation state and model the transition state. Also, we capture the consecutive conformational change from local chemical reaction to functional expression. Through the research, we will deepen the understanding of relationship between structure and function of protein molecule and try to find a concept of “functional diversity and functional conversion by evolution”.
研究図Research Figure
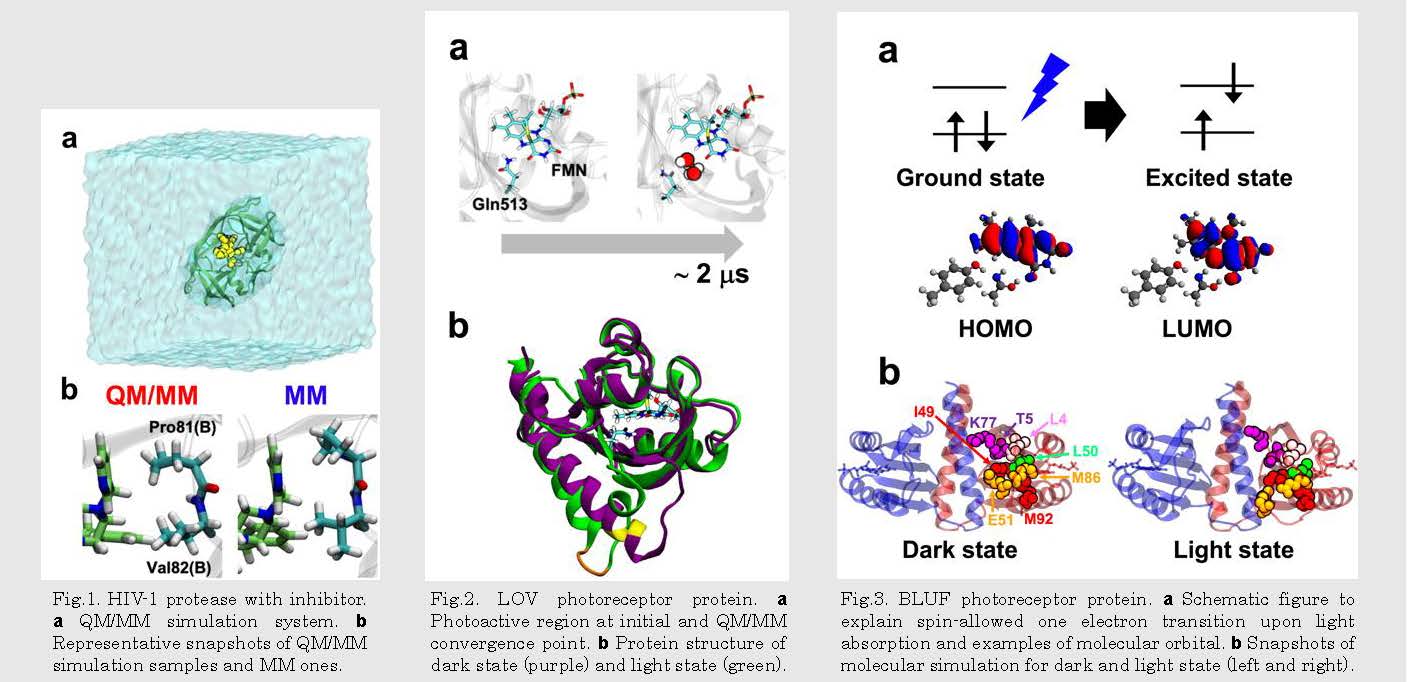
Fig.2. LOV photoreceptor protein. a Photoactive region at initial and QM/MM convergence point. b Protein structure of dark state (purple) and light state (green).
Fig.3. BLUF photoreceptor protein. a Schematic figure to explain spin-allowed one electron transition upon light absorption and examples of molecular orbital. b Snapshots of molecular simulation for dark and light state (left and right).
文献 / Publications
J. Chem. Inf. Model. 62, 1328 (2022). Curr. Opin. Struc. Biol. 71, 7 (2021). Phys. Rev. B 97, 104514 (2018). J. Phys. Soc. Japan 85, 084704 (2016). Phys. Rev. B 92, 024507 (2015).
研究者HP
- masahiko.taguchi.c4
 tohoku.ac.jp
tohoku.ac.jp - http://www2.tagen.tohoku.ac.jp/lab/nango/html/