
RIES
Research Institute for Electronic Science, Hokkaido University
北海道大学
電子科学研究所

LAST UPDATE 2017/02/25
-
研究者氏名
Researcher Name小松崎民樹 Tamiki KOMATSUZAKI
教授 Professor -
所属
Affiliation北海道大学 電子科学研究所
附属社会創造数学研究センター データ数理研究分野
Research Institute for Electronic Science, Hokkaido University
Research Center of Mathematics for Social Creativity, Molecule & Life Nonlinear Sciences Laboratory -
研究キーワード
Research Keywords一分子生物学
分子データ科学
力学系理論、非平衡統計
エネルギー地形・複雑ネットワーク
Single molecule biology
Molecular data science
Dynamical systems theory, nonequilibrium statistical mechanics
Energy landscapes・complex network
- 研究テーマ
Research Subject -
1分子時系列およびイメージング情報から読み解く生命システムの階層性・頑健性の解明
Deciphering hierarchy and robustness of living systems from single molecule time series and imaging data
研究の背景 Background of the Research
膨大な数の分子群から構成される細胞小器官、細胞などの生命システムに対して、1分子分光、分子イメージングなどによる実験研究は飛躍的な進展を遂げつつある。しかしながら、一方、ハミルトニアン、散逸力学系などに依拠する既存の分子理論ではそのモデル自体の適用が非自明であり、対応すべき理論研究が大幅に立ち遅れている。データ駆動型の新しい数理モデリング技術の創出、ならびに非平衡環境下における状態変化の「偶然と必然」の原理的理解が待望視されている。
Experimental researches such as single-molecule spectroscopy, molecular imaging are making great breakthroughs to enable us to decipher living systems such as organelles and cells consisting of an enormous number of molecules. On the other hand, it is non-trivial to adopt existing molecular theories which lean on Hamiltonian, dissipative dynamical systems and the like to these more complex systems. Research concerning the development of chemical theories for complex systems is lagging behind significantly, and the development of new data-driven chemical theories and the deep understanding of principle on chance and necessity for changes of states are eagerly awaited.
研究の目標 Research Objective
多彩なミクロ環境場における分子の動態挙動を計測する1分子計測、1分子イメージングに依拠して、その動態システムを記述する分子モデルを予め仮定するのではなく、得られる実験データから抽出する新しい分子データ科学を創出する。非線形物理、数理生物学、数理統計学、ネットワーク理論などの周辺学問領域の知見を積極的に導入し、状態変化における偶然と必然の原理的理解を目指すとともに理論と実験の協働研究を展開する。
Based on single molecule measurements and imaging techniques to measure the dynamic behavior of molecules in various micro-environmental fields, instead of assuming a molecular model that describes a dynamic system, we construct a new molecular data science in terms of the experimental data. We establish to the principle of chance and necessity for changes of state and our research bridging theory and experiments will involve knowledge of surrounding academic disciplines such as nonlinear physics, mathematical biology, mathematical statistics, and network theory.
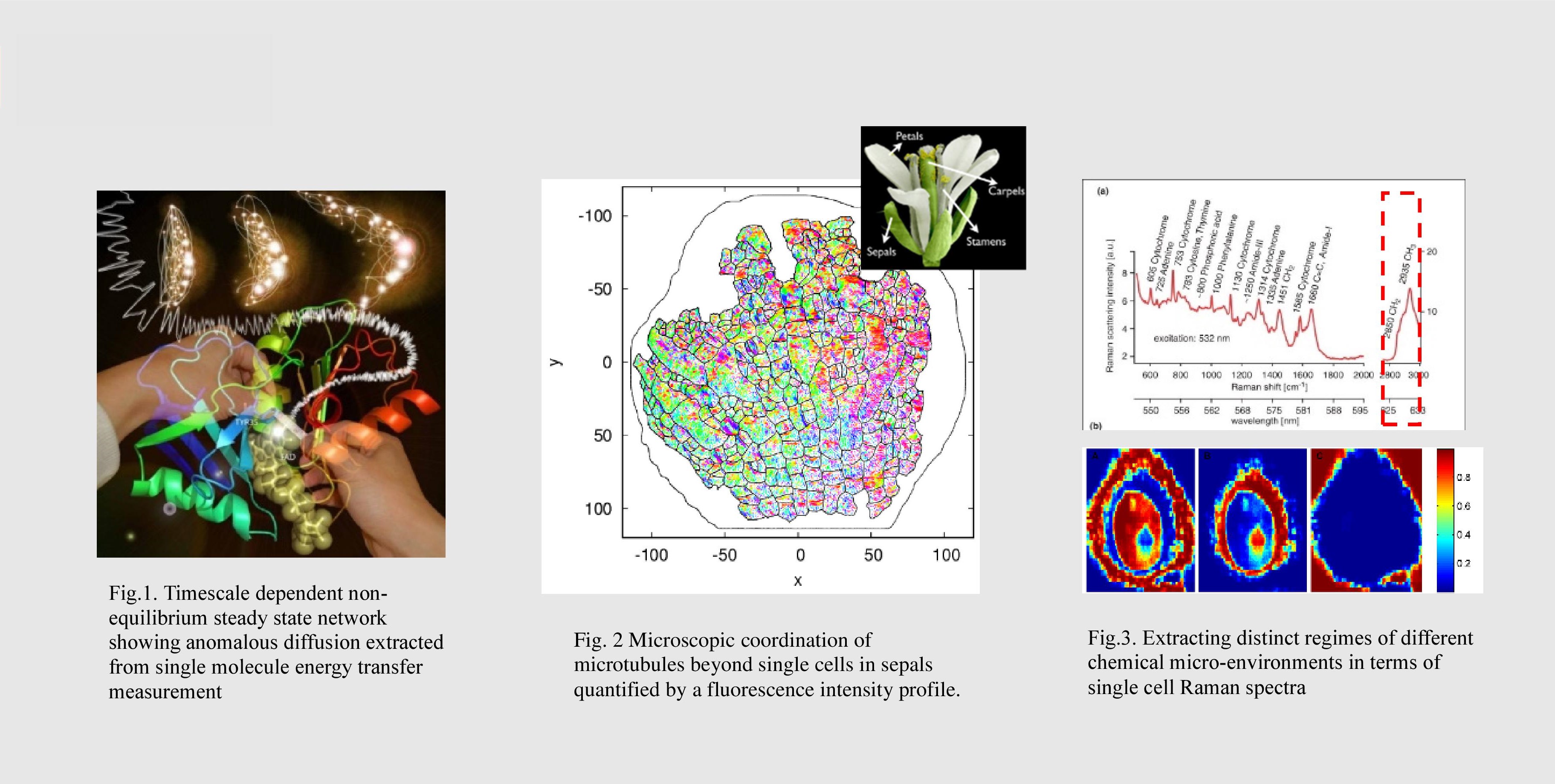
研究図Figures
論文発表 / Publications
(著書)T. Komatsuzaki他"Single Molecule Biophysics: Experiments and Theories" Adv. Chem. Phys., 146, John-Wiley & Sons, Inc (2012).
J. Phys. Chem. B (2019) in press; J. Chem. Phys. 148,123325(2018); Nature Chem. Bio. 13,1009 (2017); Developmental Cell 38,15-32 (2016); Nature Comm. 6,10223 (2015)
研究者連絡先 / HP
- tamiki
 es.hokudai.ac.jp
es.hokudai.ac.jp - http://mlns.es.hokudai.ac.jp/